Page 227 - BUAP - Facultad de Medicina - Epigenética en las patologías
P. 227
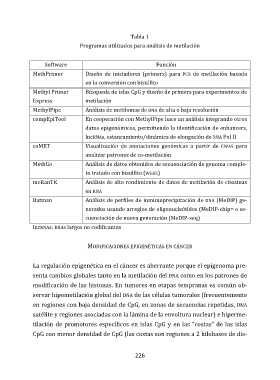
Tabla 1
Programas utilizados para análisis de metilación
Software Función
MethPrimer Diseño de iniciadores (primers) para PCR de metilación basada
en la conversión con bisulfito
Methyl Primer Búsqueda de islas CpG y diseño de primers para experimentos de
Express metilación
MethylPipe Análisis de metilomas de DNA de alta o baja resolución
compEpiTool En cooperación con MethylPipe hace un análisis integrando otros
datos epigenómicos, permitiendo la identificación de enhancers,
lncRNAs, estancamiento/dinámica de elongación de RNA Pol II
coMET Visualización de anotaciones genómicas a partir de EWAS para
analizar patrones de co-metilación
MethGo Análisis de datos obtenidos de secuenciación de genoma comple-
to tratado con bisulfito (WGBS)
meRanTK Análisis de alto rendimiento de datos de metilación de citosinas
en RNA
Batman Análisis de perfiles de inmunoprecipitación de DNA (MeDIP) ge-
nerados usando arreglos de oligonucleótidos (MeDIP-chip= o se-
cuenciación de nueva generación (MeDIP-seq)
lncRNAs: RNAs largos no codificantes
MODIFICACIONES EPIGENÉTICAS EN CÁNCER
La regulación epigenética en el cáncer es aberrante porque el epigenoma pre-
senta cambios globales tanto en la metilación del DNA como en los patrones de
modificación de las histonas. En tumores en etapas tempranas es común ob-
servar hipometilación global del DNA de las células tumorales (frecuentemente
en regiones con baja densidad de CpG, en zonas de secuencias repetidas, DNA
satélite y regiones asociadas con la lámina de la envoltura nuclear) e hiperme-
tilación de promotores específicos en islas CpG y en las “costas” de las islas
CpG con menor densidad de CpG (las costas son regiones a 2 kilobases de dis-
226